
|
|
 |
|||||||||
| |||||||||
|
Репарирующие белки скользят по ДНК
В любой клетке, будь она бактериальной или эукариотической, ДНК постоянно повреждается. Спираль ДНК рвётся, из неё выпадают фрагменты, одни нуклеотиды меняются на другие. Обильные повреждения сопровождают деление клетки, когда наследственный материал удваивается. И одновременно с репликативными молекулярными машинами в клетке постоянно работают белки систем . Таковых в клетке несколько, и одна из самых употребляемых - система пострепликативной репарации, когда необходимо восстановить повреждения уже после того, как ДНК удвоилась. Этот механизм не предполагает узнавания дефекта: он просто вырезает повреждённый участок из ДНК. Если в повреждённом месте были не нуклеотидные замены, а разрыв или пробел, белки пострепликативной репарации расширяют неправильно скопированную или повреждённую зону, чтобы проще было заменить испорченный участок. 
Суть репарации состоит в том, чтобы на место образовавшейся бреши вставить правильный фрагмент ДНК. И главный участник процесса известен давно - это белок (белок бактерий, но его гомологи есть и у эукариот). RecA работает так: он садится на ту цепь ДНК, которая осталась целой, причём много молекул белка покрывают всю цепь, оставшуюся без напарника. Получается этакая ДНК-белковая нить, которой необходимо найти комплементарный кусок среди неповреждённых нитей ДНК. Поскольку в бактериальной клетке почти постоянно идёт удвоение ДНК, целые копии определённо есть. Но вот как белок отыскивает нужную последовательность в куче других? Ведь это всё равно что искать иголку в стоге сена. По одной из теорий, молекулы RecA анализируют сразу несколько фрагментов, чтобы определить их сходство с нарушенной последовательностью, - и не так давно эта идея получила . Но, как пишут в журнале исследователи из (США), есть ещё один механизм работы RecA. Во втором случае белок вместе с нитью ДНК, на которой он сидит, начинает последовательно сканировать нити ДНК на предмет использования в качестве шаблона для залатывания повреждения.
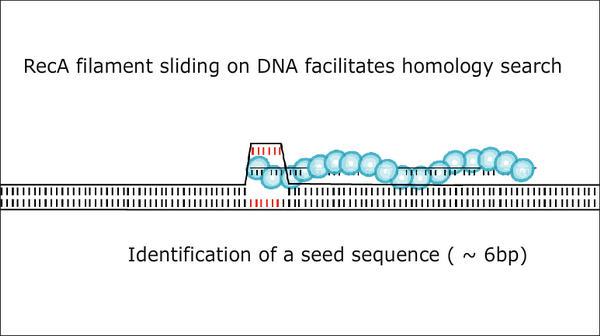
Исследователи увидели этот механизм с помощью метода, называемого фёрстеровским переносом энергии, - когда энергетический обмен между близко расположенными молекулами приводит к изменениям в свечении флюоресцентной метки. ДНК-белковая нить действительно ездила назад-вперёд по неповреждённой ДНК, и достаточно было 6-нуклеотидного совпадения между ними, чтобы запустить реакцию рекомбинации, в результате которой восстанавливался повреждённый и (или) вырезанный фрагмент. Двигаясь по ДНК-шаблону, комплекс RecA может проверить около 200 нуклеотидов. Авторы статьи утверждают, что такие скольжения ускоряют процесс репарации в 200 раз. Скорость тут является важным параметром, так как, если бы репаративная машина работала медленно, в генах быстро накапливались бы повреждения. Как соотносятся эти механизмы, одновременного перебора и скольжения, пока не ясно, но, вполне возможно, что они действуют одновременно. Репарация ДНК - такое дело, что «лишние» усилия всяко лишними не будут. Подготовлено по материалам .
|
Дизайн и поддержка: Interface Ltd. |
|